前言
目标检测主要通过人工智能技术识别并定位图像中的物体,在实际生活中应用领域广泛:数字摄像机智能火灾监控、医学影像肿瘤检测、数码相机人脸自动定位等等多个领域。传统目标检测算法因无法利用图像的深层特征而易受到物体遮挡、光照变化等因素的干扰,导致漏检与误检。深度学习的出现可以很好地解决这一问题。深度学习算法可以从样本中学习,通过对特征的加工组合,进而提取图像的更深层特征。本文在深度学习理论的支持下研究目标检测在血液细胞中的应用。。
医疗图像的获取以及标注需要耗费巨大的人力成本,而训练数据不足会导致模型出现过拟合的现象,尤其是血液细胞中各类细胞的识别。并且血液细胞图像本身存在着对比度低和各类细胞数量、外形差异较大的特点,这些都影响着血液细胞检测的精度。本文在对深度学习目标检测算法研究的基础上,对血液细胞中的各类细胞识别,并使用算法得到较高的血液细胞的检测精度。
本系统采用登录注册进行用户管理,对于图片、视频和摄像头捕获的实时画面,可检测医疗图像,系统支持结果记录、展示和保存,每次检测的结果记录在表格中。对此这里给出博主设计的界面,同款的简约风,功能也可以满足图片、视频和摄像头的识别检测,希望大家可以喜欢,初始界面如下图:
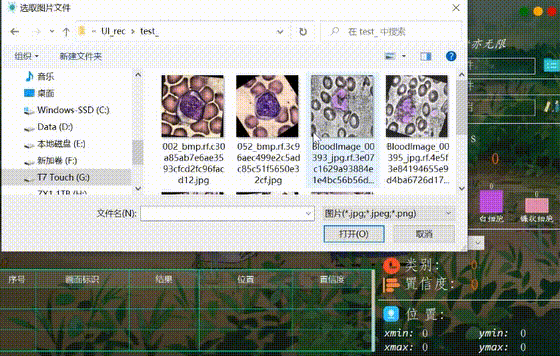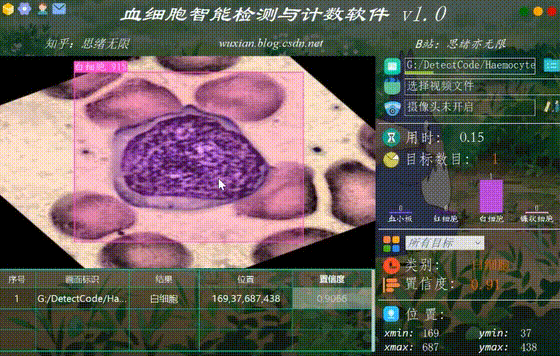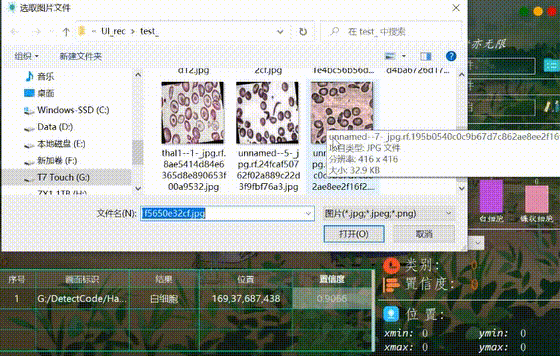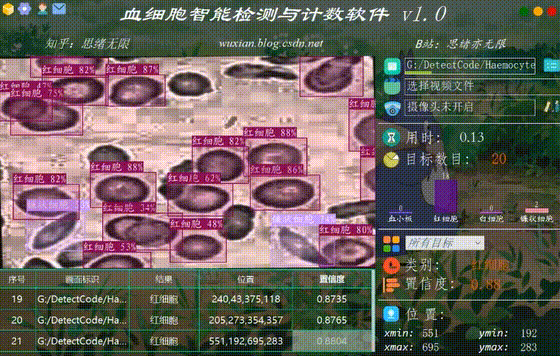
检测类别时的界面截图(点击图片可放大)如下图,可识别画面中存在的多个类别,也可开启摄像头或视频检测:
详细的功能演示效果参见博主的B站视频或下一节的动图演示,觉得不错的朋友敬请点赞、关注加收藏!系统UI界面的设计工作量较大,界面美化更需仔细雕琢,大家有任何建议或意见和可在下方评论交流。
1. 效果演示
软件好不好用,颜值很重要,首先我们还是通过动图看一下识别的效果,系统主要实现的功能是对图片、视频和摄像头画面中的血细胞进行识别,识别的结果可视化显示在界面和图像中,另外提供多个目标的显示选择功能,演示效果如下。
(一)系统介绍
血细胞智能检测与计数软件主要用于显微镜下的血细胞检测与计数,基于深度学习技术识别图像中常见的4种血细胞,包括血小板、红细胞、白细胞、镰状细胞等,输出细胞的标记框坐标和类别,以辅助自动化细胞统计和医学研究;软件提供登录注册功能,可进行用户管理;软件能够有效识别电子显微镜采集的细胞图片、视频等文件形式,检测各种细胞形态,并记录识别结果在界面表格中方便查看;可开启摄像头实时监测和统计当前视野范围各种类型细胞数目,支持结果记录、展示和保存。
(二)技术特点
(1)YoloV5目标检测算法检测血细胞,模型支持更换;
(2)摄像头实时检测血细胞,展示、记录和保存结果;
(3)检测图片、视频等图像中的血细胞个体;
(4)支持用户登录、注册,检测结果可视化功能;
(三)用户注册登录界面
这里设计了一个登录界面,可以注册账号和密码,然后进行登录。界面还是参考了当前流行的UI设计,左侧是一个动图,右侧输入账号、密码、验证码等等。
(四)选择图片识别
系统允许选择图片文件进行识别,点击图片选择按钮图标选择图片后,显示所有识别的结果,可通过下拉选框查看单个结果,以便具体判断某一特定目标。本功能的界面展示如下图所示:
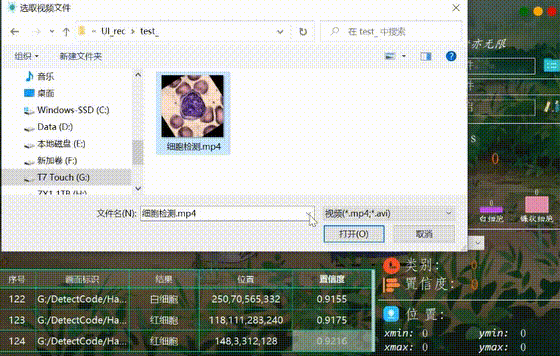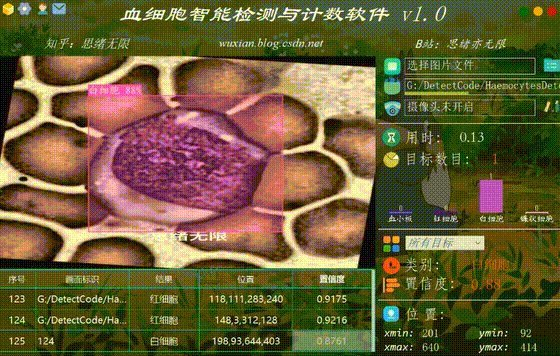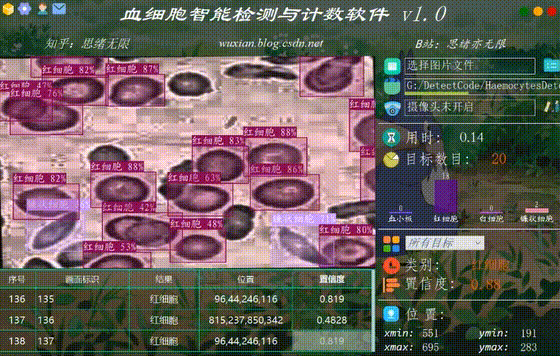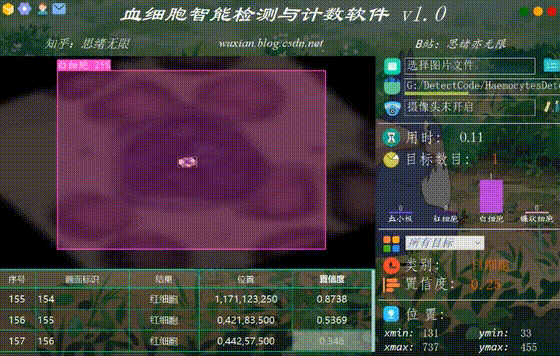
(五)视频识别效果展示
很多时候我们需要识别一段视频中的血细胞,这里设计了视频选择功能。点击视频按钮可选择待检测的视频,系统会自动解析视频逐帧识别多个血细胞,并将血细胞的分类和计数结果记录在右下角表格中,效果如下图所示:
(六)摄像头检测效果展示
在真实场景中,我们往往利用摄像头获取实时画面,同时需要对血细胞进行识别,因此本文考虑到此项功能。如下图所示,点击摄像头按钮后系统进入准备状态,系统显示实时画面并开始检测画面中的血细胞,识别结果展示如下图:
2. 血细胞数据集和模型训练
(一)数据集制作
本文实验的血细胞数据集包含训练集2853张图片,验证集219张图片,测试集81张图片,共计3153张图片,选取部分数据部分样本数据集如图所示。
每张图像均提供了图像类标记信息,图像中细胞的bounding box,目标的属性信息,数据集并解压后得到如下的图片
船舰数据集的类别信息如下,包含血小板、红细胞、白细胞、镰状细胞等类别
python Chinese_name = {'Platelets': "血小板", 'RBC': "红细胞", 'WBC': "白细胞", 'sickle cell': "镰状细胞"}
原数据格式是xml文件对目标细胞注释,现在需要将这种注释转换为yolov5所需的格式。即每个图像对应一个txt文件,文件中存储该图像中全部细胞的类别和坐标,一行存储一个细胞的信息,如下图
在本项目的同级目录下创建数据集存储路径,路径内创建训练集,测试集路径和配置文件dataset.yaml
images内存储图像数据,labels存储标注数据,文件名称对应相同,设置数据集配置文件如下
python train: ./Haemocytes/images/train val: ./Haemocytes/images/valid test: ./Haemocytes/images/test nc: 4 names: ['Platelets', 'RBC', 'WBC', 'sickle cell']
(1)train 指定训练集图像路径
(2)val 指定验证集图像路径
(3)nc 指定目标类别数量 这里为血小板,红细胞,白细胞共3种
(4)目标对应类别名称
接下来需要配置项目路径内的train.py的相关参数,指定预训练的模型权重和模型结构文件和数据集配置文件,训练的轮数,batch_size的大小和输入图像的分辨率。
python parser = argparse.ArgumentParser() parser.add_argument('--weights', type=str, default='yolov5s.pt', help='initial weights path') parser.add_argument('--cfg', type=str, default='models/yolov5s.yaml', help='model.yaml path') parser.add_argument('--data', type=str, default='dataset.yaml', help='data.yaml path') parser.add_argument('--hyp', type=str, default='data/hyp.scratch.yaml', help='hyperparameters path') parser.add_argument('--epochs', type=int, default=300) parser.add_argument('--batch-size', type=int, default=1, help='total batch size for all GPUs') parser.add_argument('--img-size', nargs='+', type=int, default=[640, 640], help='[train, test] image sizes')
在我们的训练过程中,mAP50作为一种常用的目标检测评估指标很快达到了较高水平,而mAP50:95也在训练的过程中不断提升,说明我们模型从训练-验证的角度表现良好。读入一个测试文件夹进行预测,通过训练得到的选取验证集上效果最好的权重best.pt进行实验,得到PR曲线如下图所示。
在深度学习中,我们通常通过损失函数下降的曲线来观察模型训练的情况。而YOLOv5训练时主要包含三个方面的损失:矩形框损失(box_loss)、置信度损失(obj_loss)和分类损失(cls_loss),在训练结束后,我们也可以在logs目录下找到生成对若干训练过程统计图。下图为博主训练血细胞识别的模型训练曲线图。
以PR-curve为例,可以看到我们的模型在验证集上的均值平均准确率为0.794。
3. 血细胞检测识别
运行testVideo得到预测结果,我们便可以将帧图像中的血细胞框出,然后在图片上用opencv绘图操作,输出血细胞的类别及血细胞的预测分数。以下是读取血细胞视频并进行检测的脚本,首先将图片数据进行预处理后送predict进行检测,然后计算标记框的位置并在图中标注出来。
python while True: # 从视频文件中逐帧读取画面 (grabbed, image) = vs.read() # 若grabbed为空,表示视频到达最后一帧,退出 if not grabbed: print("[INFO] 运行结束...") output_video.release() vs.release() exit() # 获取画面长宽 if W is None or H is None: (H, W) = image.shape[:2] image = cv2.resize(image, (850, 500)) img0 = image.copy() img = letterbox(img0, new_shape=imgsz)[0] img = np.stack(img, 0) img = img[:, :, ::-1].transpose(2, 0, 1) # BGR to RGB, to 3x416x416 img = np.ascontiguousarray(img) pred, useTime = predict(img) det = pred[0] p, s, im0 = None, '', img0 if det is not None and len(det): # 如果有检测信息则进入 det[:, :4] = scale_coords(img.shape[1:], det[:, :4], im0.shape).round() # 把图像缩放至im0的尺寸 number_i = 0 # 类别预编号 detInfo = [] for *xyxy, conf, cls in reversed(det): # 遍历检测信息 c1, c2 = (int(xyxy[0]), int(xyxy[1])), (int(xyxy[2]), int(xyxy[3])) # 将检测信息添加到字典中 detInfo.append([names[int(cls)], [c1[0], c1[1], c2[0], c2[1]], '%.2f' % conf]) number_i += 1 # 编号数+1 label = '%s %.2f' % (names[int(cls)], conf) # 画出检测到的目标物 plot_one_box(image, xyxy, label=label, color=colors[int(cls)]) # 实时显示检测画面 cv2.imshow('Stream', image) image = cv2.resize(image, (vw, vh)) output_video.write(image) # 保存标记后的视频 if cv2.waitKey(1) & 0xFF == ord('q'): break # print("FPS:{}".format(int(0.6/(end-start)))) frameIndex += 1
执行得到的结果如下图所示,图中血细胞的种类和置信度值都标注出来了,预测速度较快。基于此模型我们可以将其设计成一个带有界面的系统,在界面上选择图片、视频或摄像头然后调用模型进行检测。
博主对整个系统进行了详细测试,最终开发出一版流畅得到清新界面,就是博文演示部分的展示,完整的UI界面、测试图片视频、代码文件,以及Python离线依赖包(方便安装运行,也可自行配置环境),均已打包上传,感兴趣的朋友可以通过下载链接获取。