引言
本系列讲解 空间转录组学 (Spatial Transcriptomics) 相关基础知识与数据分析教程,持续更新,欢迎关注,转发,文末有交流群!
QC 质量控制
我们使用 SpotSweeper 对子集化的 16 µm 数据进行质量控制。
# calculate per-cell QC metrics
mt <- grepl("^MT-", rownames(.vhd16))
.vhd16 <- addPerCellQCMetrics(.vhd16, subsets=list(mt=mt))
# determine outliers based on
# - low log-library size
# - few uniquely detected features
# - high mitochondrial count fraction
.vhd16 <- localOutliers(.vhd16, metric="sum", direction="lower", log=TRUE)
.vhd16 <- localOutliers(.vhd16, metric="detected", direction="lower", log=TRUE)
.vhd16 <- localOutliers(.vhd16, metric="subsets_mt_percent", direction="higher", log=TRUE)
.vhd16$discard <-
.vhd16$sum_outliers |
.vhd16$detected_outliers |
.vhd16$subsets_mt_percent_outliers
# tabulate number of bins retained
# vs. removed by any criterion
table(.vhd16$discard)
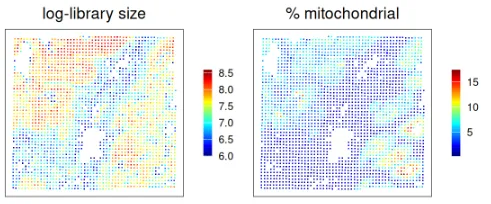
我们可以将 SpotSweeper 在空间中识别的局部异常值可视化:
plotCoords(.vhd16, point_shape=15, annotate="discard") + ggtitle("discard") +
plotCoords(.vhd16, point_shape=15, annotate="sum_outliers") + ggtitle("low_lib_size") +
plotCoords(.vhd16, point_shape=15, annotate="detected_outliers") + ggtitle("low_n_features") +
plot_layout(nrow=1, guides="collect") &
theme(
plot.title=element_text(hjust=0.5),
legend.key.size=unit(0, "lines")) &
guides(col=guide_legend(override.aes=list(size=3))) &
scale_color_manual("discard", values=c("lavender", "purple"))
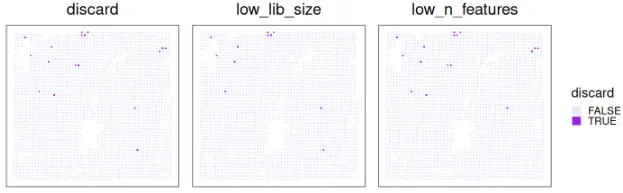
最后,我们将 Visium HD 16 µm 对象子集到通过 QC 的容器中:
.vhd16 <- .vhd16[, !.vhd16$discard]
dim(.vhd16)
聚类
首先,我们确定目标区域中的高变异基因(HVG):
.vhd16 <- logNormCounts(.vhd16)
dec <- modelGeneVar(.vhd16)
hvg <- getTopHVGs(dec, n=3e3)
首先通过一对空间核函数捕捉基因表达在空间上的变化,接着进行降维处理,再通过图聚类方法划分出不同的空间区域。
# set seed for random number generation
# in order to make results reproducible
set.seed(112358)
# 'Banksy' parameter settings
k <- 8 # consider first order neighbors
l <- 0.2 # use little spatial information
a <- "logcounts"
xy <- c("array_row", "array_col")
# restrict to selected features
tmp <- .vhd16[hvg, ]
# compute spatially aware 'Banksy' PCs
tmp <- computeBanksy(tmp, assay_name=a, coord_names=xy, k_geom=k)
tmp <- runBanksyPCA(tmp, lambda=l, npcs=20)
reducedDim(.vhd16, "PCA") <- reducedDim(tmp)
## run UMAP (for visualization purposes only)
# .vhd16 <- runUMAP(.vhd16, dimred="PCA")
# build cellular shared nearest-neighbor (SNN) graph
g <- buildSNNGraph(.vhd16, use.dimred="PCA", type="jaccard", k=20)
# cluster using Leiden community detection algorithm
k <- cluster_leiden(g, objective_function="modularity", resolution=1.2)
table(.vhd16$Banksy <- factor(k$membership))
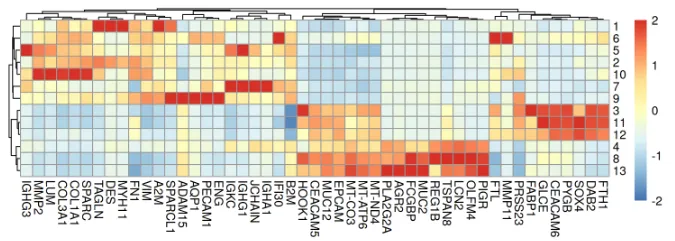
接下来,我们可以进行差异基因表达(DGE)分析,以识别每个聚类的标记基因;我们还计算所选标记基因在各聚类中的平均表达水平:
# differential gene expression analysis
mgs <- findMarkers(.vhd16, groups=.vhd16$Banksy, direction="up")
# select for a few markers per cluster
top <- lapply(mgs, \(df) rownames(df)[df$Top <= 2])
top <- unique(unlist(top))
# average expression by clusters
pbs <- aggregateAcrossCells(.vhd16,
ids=.vhd16$Banksy, subset.row=top,
use.assay.type="logcounts", statistics="mean")
每个簇中的标记基因可以通过热图可视化:
# visualize averages z-scaled across clusters
pheatmap(
mat=t(assay(pbs)), scale="column", breaks=seq(-2, 2, length=101),
cellwidth=10, cellheight=10, treeheight_row=5, treeheight_col=5)
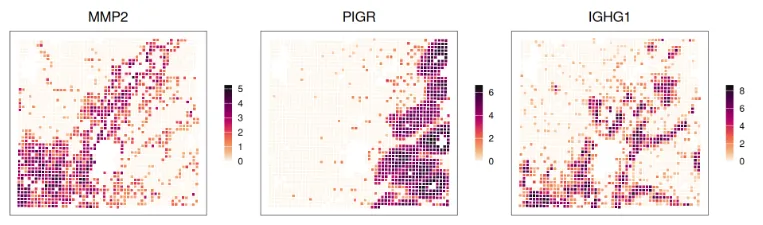
或者,我们可以可视化空间中选定标记的 bin-wise 表达:
gs <- c("MMP2", "PIGR", "IGHG1")
ps <- lapply(gs, \(.) plotCoords(.vhd16, annotate=., point_shape=15,
point_size=0.8, assay_name="logcounts"))
wrap_plots(ps, nrow=1) & theme(
legend.key.width=unit(0.5, "lines"),
legend.key.height=unit(1, "lines")) &
scale_color_gradientn(colors=rev(hcl.colors(9, "Rocket")))